Génomique intégrative du Neurodéveloppement
Le groupe Génomique intégrative du neurodéveloppement étudie le développement cérébral normal et pathologique par le biais d’approches de biologie statistique et computationnelle. Nous nous intéressons particulièrement aux troubles du neurodéveloppement et aux maladies neuropsychiatriques dont l’épilepsie, les troubles bipolaires et les complications de la prématurité.
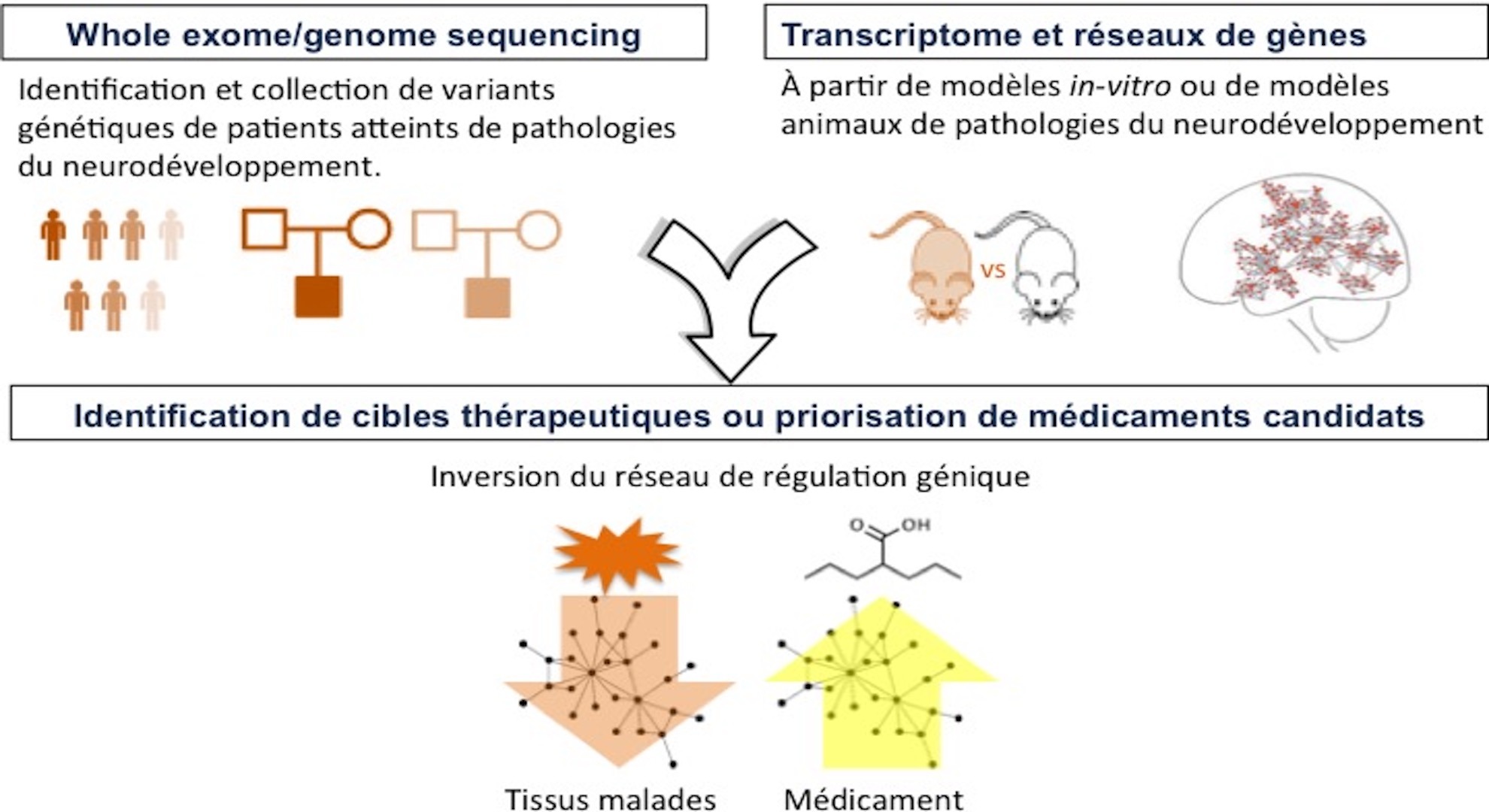
Nous développons des outils pour analyser et combiner des jeux de données en accès libre, ou propres au laboratoire ou à nos collaborateurs extérieurs. Nous exploitons des données issues de modèles animaux, de modèles in vitroainsi que de patients, et d’individus sains. L’étude du transcriptome de tissus et de cellules permet de révéler des régulateurs influençant une maladie étudiée. Nous nous intéressons particulièrement aux réseaux de régulation génique et aux méthodes d’analyse de réseaux pour prioriser les régulateurs clés qui régissent les processus développementaux et pathologiques. Une autre stratégie que nous utilisons pour l’étude des troubles de l’humeur consiste à intégrer des données multi-omiques aux données phénotypiques des patients pour identifier des biomarqueurs candidats, et aider à stratifier des groupes de patients. Au-delà d’une meilleure compréhension des mécanismes génétiques et moléculaires du neurodéveloppement et des troubles neuropsychiatriques, notre but est d’identifier de nouvelles cibles thérapeutiques et d’orienter vers de nouveaux usages de médicaments existants. Nous étudions en particulier l’utilisation de méthodes d’apprentissage statistique (“machine learning”) pour repositionner les médicaments en fonction de leur impact transcriptionnel.
Le criblage virtuel basé sur la structure (SBVS) vise à identifier des composés bioactifs avec des approches informatiques en utilisant comme point de départ la structure 3D de la cible protéique. Ce processus implique le docking et le classement d’un grand nombre de molécules en fonction de scores de liaison prédits, suivis d’une sélection interactive d’une liste restreinte de molécules pour des tests expérimentaux. Afin de faciliter la prise de décision, les propriétés ADMET sont également prédites. Notre groupe se spécialise dans la conception d’inhibiteurs d’interactions protéine-protéine, en utilisant et en développant des approches telles que le docking combiné à l’apprentissage automatique pour optimiser la découverte de composés prometteurs et de candidats-médicaments.
La bioinformatique structurale joue un rôle clé dans l’étude de l’impact des mutations ponctuelles sur la structure et la fonction des protéines. Ces outils tentent de prédire comment des mutations peuvent altérer la structure 3D, la stabilité et les interactions d’une protéine avec d’autres molécules. Les simulations, le docking et les calculs d’énergie permettent d’évaluer les modifications de l’affinité de liaison, de l’activité enzymatique ou du repliement, fournissant des informations sur les mécanismes moléculaires sous-jacents. Associées à des études expérimentales, ces approches facilitent la compréhension des relations entre génotype et phénotype.
Contacts
Andrée Delahaye-Duriez
Inserm UMR 1141
Hôpital robert Debré
48 boulevard Sérurier
75019 Paris
andree.delahaye@inserm.fr
Bruno Villoutreix
Inserm UMR 1141
Hôpital robert Debré
48 boulevard Sérurier
75019 Paris
bruno.villoutreix@inserm.fr
À lire aussi

Un nouvel article Organoïdes en 2026 publié dans iScience
https://www.cell.com/iscience/fulltext/S2589-0042(26)00332-9

Séminaire scientifique d’Alexandra Benchoua le jeudi 16 Avril 2026 (11h) : Modèles de cellules souches pluripotentes humaines pour le criblage à haut débit de molécules thérapeutiques et la médecine de précision dans les TND.
Salle de réunion 6ème étage du bâtiment Bingen et Visio - https://u-paris.zoom.us/j/84730677369?pwd=AFdhdI6alNWLbXub7sM1DJ33yUIOYN.1 - Résumé 20260416 séminaire A-Benchoua

Séminaire scientifique Dr. Clément Apelian le jeudi 19 février 2026 (11h).
Nous aurons le plaisir d’accueillir le Dr. Clément Apelian du laboratoire Laboratoire PRHySME pour un séminaire intitulé: L'hypnose au-delà du fantasme - défis fondamentaux et intérêt clinique 20260219 séminaire Clément Alepian

Séminaire scientifique Pr. Raul Chavez-Valdez le jeudi 12 février 2026 (14h).
Nous aurons le plaisir d’accueillir le Pr. Raul Chavez-Valdez de l'université de Baltimore pour un séminaire intitulé: Synaptic Plasticity following Neonatal Hypoxic-Ischemic Brain Injury 20260212 séminaire Raul Chavez english ...
